Docker en mer
Publié le jeudi 24 novembre 2016.Dernière mise à jour : 18/09/2017
Introduction
Lors d’une campagne océanographique, l’équipe scientifique met en œuvre sur les navires de recherche ses propres équipements de collecte de données. Ces navires sont également équipés d’instruments mesurant de nombreux paramètres le long de la route. Il est important, voir primordial, de vérifier en temps quasi réel la qualité des données acquises afin de palier rapidement à tout dysfonctionnement.
Pour cela, il faut extraire les données acquises dans des formats hétérogènes et les transformer dans des formats"standards" comme le NetCDF, ASCII et/ou XML, formats à partir desquels nous pourrons visualiser les données avec des outils génériques comme dataGUI fonctionnant sous Matlab, Ferret ou ODV.
Depuis plusieurs années, j’ai mis en place un Système d’Information (SI) facilement transposable sur l’ensemble des navires de la flotte nationale.
Les données sont collectées dans 2 arborescences de répertoires depuis un partage réseau accessible via samba sous Windows ou NFS pour Linux. Le premier répertoire, nommé "data-raw", contient les données brutes, avec un sous répertoire par instrument de mesure. Le second, "data-processing", contient les données traitées en temps réel, données qui peuvent être corrigées, ainsi que les scripts de traitements associés. Le système d’information contiendra d’autres répertoires comme "data-cal", "data-adjusted" et "data-final" qui seront utilisés généralement après la campagnes, respectivement pour la calibration, l’ajustement et la diffusion des données. Nous trouvons dans notre SI un cinquième répertoire, baptisé "local" qui contiend les scripts et programmes utilisés pendant la campagne. Sous local/etc/skel on trouvera un script shell d’initialisation des alias et des fonctions d’automatisation des traitements.
Actuellement, la quasi totalité des traitements est réalisée par des scripts Perl. Nous avons un script générique par instrument. La configuration de l’ensemble des scripts est définie dans un unique fichier de configuration (.ini) que l’on retrouvera à la racine du répertoire "data-processing".
Le rôle des scripts consiste à lire l’ensemble des fichiers de données présents dans les sous répertoire data, d’extraire les entêtes : date, position, etc ...puis les mesures, qui peuvent être de type profil, trajectoire ou série temporelle. En sortie, nous obtenons un fichier « d’entête », un fichier au format ASCII, un fichier au format XML qui contiendra en plus les méta-data ainsi qu’un fichier au format ODV (Ocean Data View). Le fichier XML est ensuite convertit en NetCDF OceanSITES avec un script Perl de transformation générique.
Un description complète du SI est détaillée dans le document suivant :
Instruction décrivant le Système d’Information, les méthodes d’acquisition et de traitement des données en mer et au laboratoire
Je travaille actuellement sur un programme écrit en langage Go, multiplateforme, qui réalise la lecture et la transformation à la volée dans les différents formats de fichiers pour tous les type d’instrument. La configuration est décrite dans un seul fichier de configuration au format TOML. Ce programme est déjà fonctionnel pour les données des sondes CTD Seabird, voir le projet oceano2oceansitessous Github. Ce programme devrait à terme remplacer les scripts Perl avec l’avantage de pouvoir être utilisé en natif sur le PC d’acquisition.
L’acquisition des données des instruments que nous utilisons est réalisée en majorité avec des logiciel sous Windows. Lorsque j’ai commencé à la fin des années 90 à mettre en place ces traitements, ils étaient réalisés sous Solaris (SUN). L’époque, les navires avaient tous au moins un serveur de ce type en libre service, mais pour retrouver un environnement de développement optimal, il était nécessaire de recompiler dans son répertoire utilisateur une bonne partie des logiciels GNU, le tout sans accès à internet, ni mail !
J’ai ensuite utilisé pendant plusieurs années Cygwin, voir le billet sur le blog de l’unité de service IMAGO, mais son déploiement n’est pas toujours facile et la mise à jour des paquets logiciels m’a bien souvent posée beaucoup de soucis, comme celle des modules Perl PDL et NetCDF. De plus, il m’arrive souvent d’intervenir lors de campagne avec des systèmes d’acquisition appartenant à d’autres laboratoires. Il n’est pas toujours possible d’installer ses propres outils sur des PC qui ne nous appartiennent pas.
A la fin des années 2000, j’ai commencé à utiliser des machines virtuelles, VMware puis VirtualBox. Solutions plus simple à déployer, il suffit d’installer le logiciel de virtualisation sur le PC hôte et de préparer une image Linux du système invité. La mise en place de l’image nécessitant tout de même une phase d’installation et d’administration plus ou moins longue.
Finalement, ces dernières années, j’ai utilisé des mini PC (NUC) sous Linux et même un Rasperry PI pour réaliser la collecte et le traitement automatique des données. Comme nous avons des liaisons Vsat depuis peu sur l’ensemble des navires, il est possible de se connecter en SSH sur ces machines via un VPN et une machine rebond, afin de déployer l’arborescence du SI et préparer la configuration des scripts ainsi que le traitement des données. Il est également possible de vérifier à distance le bon fonctionnement des traitements, ce qui est un réel progrès.
Bon, et bien qu’en est il de Docker dans tout cela ?
En y réfléchissant un peu, il y a 2 bonnes raisons de tester l’utilisation de Docker dans notre contexte de travail :
- Créer des images contenant l’ensemble des logiciels nécessaires aux traitements des données, images que l’on pourra faire évoluer et dupliquer facilement d’un système à un autre
- Déployer un container Docker sous Linux et l’utiliser sous Windows afin de faire cohabiter sur une même machine les logiciels d’acquisition et de traitement
- Faciliter le déploiement et la maintenance de son environnement de travail
Je ne ferais pas une description détaillée de Docker ici, le net foisonne de littérature sur le sujet. Pour installer Docker, aller sur la page Docker for Windows.
Sous Windows 7 et 8, il faut installer Docker Toolbox qui va installer les composants suivants :
- Virtualbox : hyperviseur permettant d’exécuter une machine virtuelle (VM).
- boot2docker : image ISO légère de Linux lancé avec VirtualBox.
- docker.exe : client docker fonctionnant sous Windows
- docker-machine.exe : logiciel communiquant avec Virtualbox pour configuer et gérer la machine virtuelle Linux.
- Kitematic : logiciel de gestion des images Docker (GUI)
- Quickstart terminal : terminal Windows sous bash permettant d’utiliser les images Docker.
Remarque : Il est fortement conseillé d’installer le logiciel ConEmu qui offre toutes les fonctionnalités d’une console Linux sous Windows et intègre l’utilisation de Docker.
Attention : Kitematic ne fonctionne pas si Gow (Gnu On Windows) est installé sur votre PC.
Gow : c’est un ensemble d’utilitaires Unix fonctionnant sous Windows.
Sous Windows 10, Docker fonctionne en natif. Il est donc possible de faire tourner dans un Powershell Windows un conteneur créé depuis une machine Linux et de le mettre à jour via le Docker hub avec un ensembles d’outils offrants des fonctionnalités comparables à celle de Git.
Voici le Dockerfile utilisé pour générer l’image du conteneur sous Github :
https://github.com/jgrelet/docker-oceano
ainsi que le dépôt de l’image sous Docker Hub :
https://hub.docker.com/r/jgrelet/perl/
Lancement de ConEmu puis sélectionner le menu Tools::Docker :
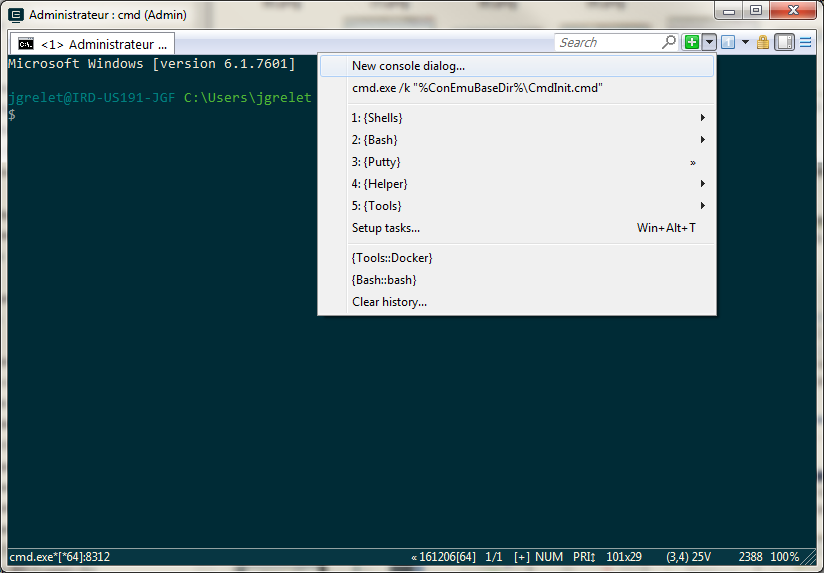
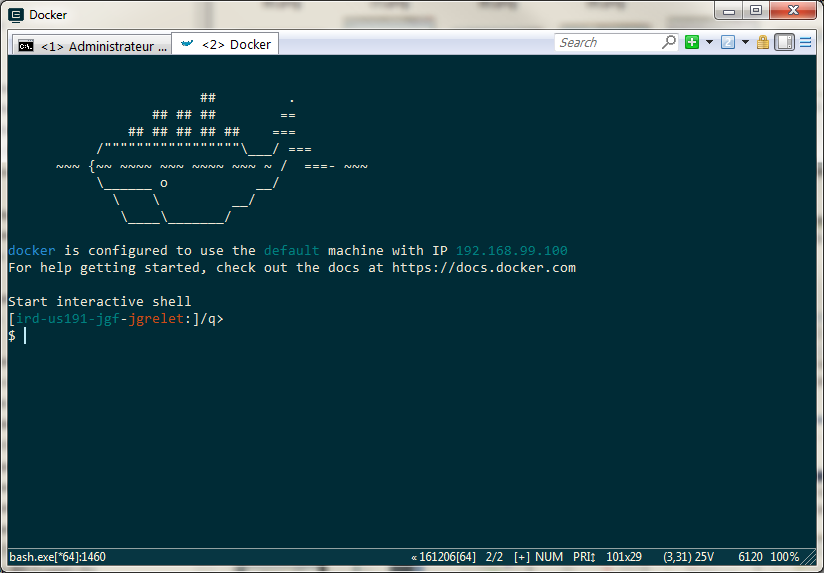
Si Docker est correctement installé sur votre machine, vous devriez obtenir l’écran suivant :
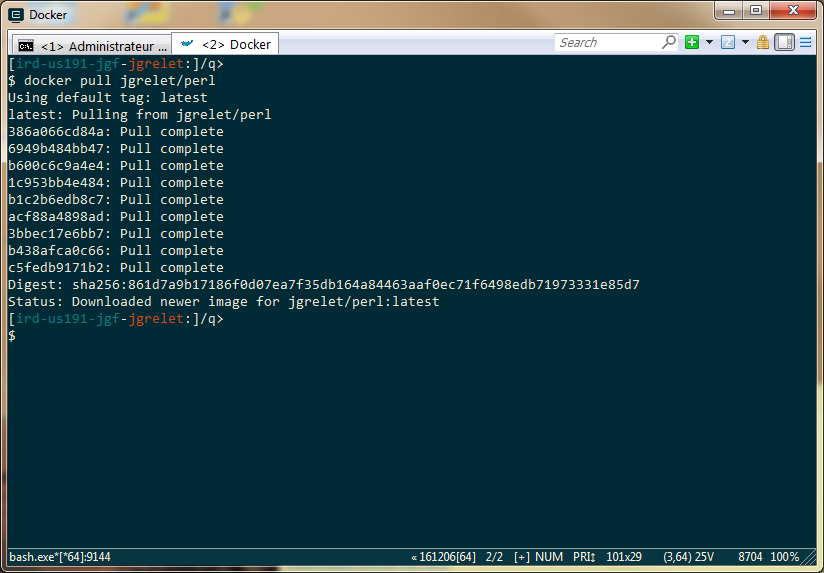
Récupérer l’image sur le Docker Hub avec la commande docker pull :
> docker pull jgrelet/perl
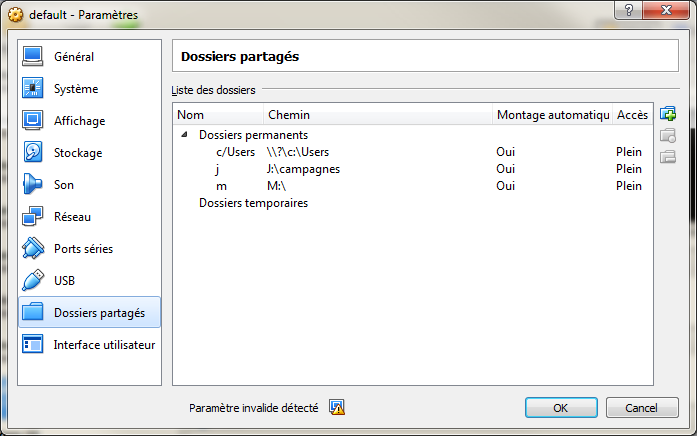
Une fois l’image téléchargée, il est nécessaire que le conteneur puisse accéder aux données. Pour cela il faut configurer l’accès aux dossiers partagés sous VirtualBox. Ici, j’ai ajouté 2 dossiers partagés :
- j pour les données de campagnes sur un disque USB externe
- m pour les données de campagnes accessibles sur un partage réseau smb
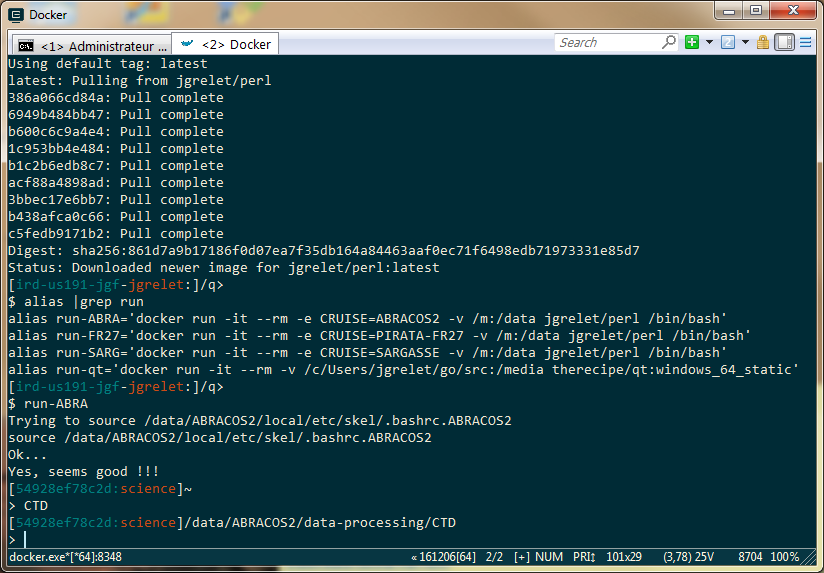
Pour lancer le conteneur, taper la commande suivante :
> docker run -it --rm -e CRUISE=ABRACOS2 -v /m:/data jgrelet/perl /bin/bashLe conteneur est lancé avec les arguments suivants :
- -it : en mode interactif
- —rm : le conteneur sera supprimé de la mémoire
- -e : la variable d’environnement CRUISE permet de se placer dans l’arborescence du Système d’Information utilisé pour nos campagnes.
- -v : le dossier partagés /m est monté au lancement du conteneur
Sous Windows 10, il faut autoriser le partage du disque réseau ou externe pour qu’il soit disponible pour le conteneur :
clic droit sur l’icône Docker de la barre des taches -> settings -> shared drives
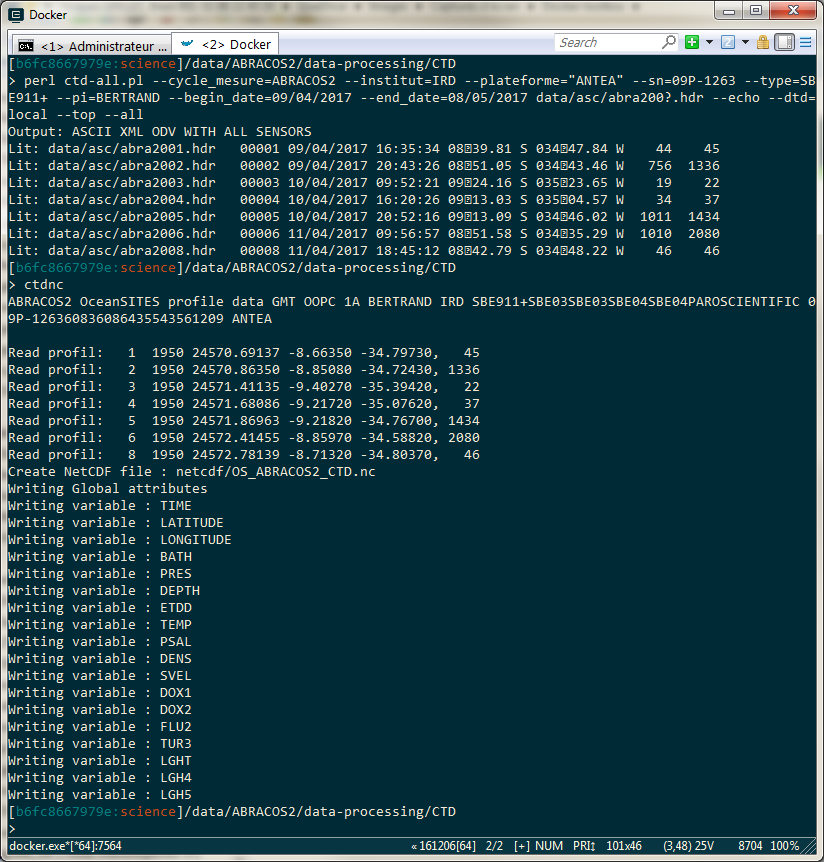
En exemple, on lance ci-dessous le traitement des 10 premières stations CTD de la campagne ABRACOS2 ainsi que la génération du fichier NetCDF, exactement comme si on était sur une machine Linux en natif :
Problèmes rencontrés
Lorsque j’ai écrit mon premier Dockerfile, je me suis basé sur une image officielle Perl du Docker Hub, image déployant la dernière version 5.24.0 de Perl.
Après installation des paquets Perl nécessaires pour l’application, impossible de lancer les scripts Perl utilisant les modules installés, le Dynaloader, qui permet de charger dynamiquement des librairies C, ne trouvaient pas c’est librairies, faisant référence à des chemins pointant vers une version 5.20.2 alors que la variable Perl @INC pointait elle vers la version installée, soit 5.24.0
> perl -le"print for @INC"
/usr/local/share/perl/5.24.0
...J’ai essayé d’installer les modules Perl avec cpanminus depuis les sources en modifiant le Dockerfile sans plus de succès :
# Install perl modules
RUN apt-get install -y cpanminus
RUN cpanm XML::LibXML \
...J’ai donc modifié mon Dockerfile pour utiliser une distribution Debian sans la couche Perl, et la surprise, Docker me dit que que le conteneur est déjà installé, et bingo, en le lançant avec docker run, Perl y est déjà installé, et avec la version 5.20.2 !
En utilisant l’image debian dans la directive FROM du Dockerfile, l’ensemble des scripts fonctionnent correctement.
Sous Windows 7, la version de Docker Toolbox 1.13 ne fonctionne pas correctement pour le montage des volumes. Par contre, pas de problème avec la précédente version 1.12 ainsi qu’avec la dernière version baptisée 17 CE pour Community Edition.
Conclusion :
Pour l’instant, je n’ai pu déployer et tester le conteneur sous Windows 10 que pendant quelques heures à partir d’une machine prêtée pour l’occasion. J’ai tout de même noté un temps de latence important dans l’exécution des scripts par rapport à ceux exécutés depuis un conteneur sous Linux. La version Docker Windows est semble t’il souvent mise à jour et il faudra vérifier avec les prochaines versions si le problème persiste.
L’utilisation de Docker permet d’obtenir rapidement et facilement la puissance des outils de traitement Linux sous Windows. Par contre, vu la taille de l’image utilisée (415 Mo), il faudra penser à l’installer sur le PC de traitement avant le départ en mer car son téléchargement risque d’être problématique, même avec une Vsat à bord pour l’accès à internet.
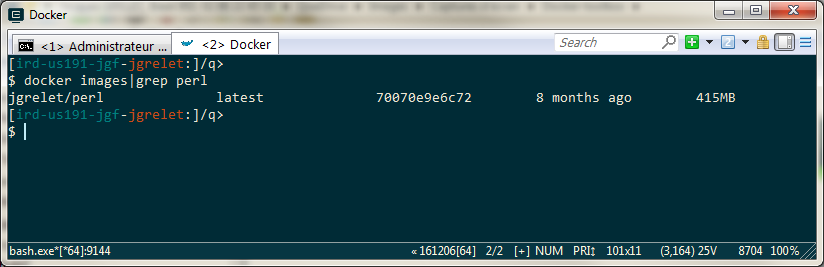
Comme alternative il faudrait utiliser une image Perl Alpine comme base de départ, à tester et configurer...
En terme de puissance de calcul, on est loin d’avoir les même performances que sous une machine Linux dédiée. Par rapport à un NUC Intel Core I5, j’ai pu noter des temps de traitements 10 fois moins rapide, ce qui lors d’une campagne avec une centaine de stations jusqu’au fond, pourrait commencer à être problématique.
Liens utiles
- En français

